2024年9月17日
OS: ubuntu 22.04
Slicer version 5.7.0
3D Slicerで長さを測りたい, 角度やROIを用いた面積・体積を測りたい場合などMarkupsモジュールを使うことで可能となる. Markupsは3D Slicerの標準機能なので追加のインストールは必要ない. Markupsモジュールはポイントを設定したり, ラインを描いたりすることに使われるが, 単にそれだけなく計測をすることも可能になっている.
メニューのModuleのプルダウンをMarkupsに合わせるとしたのような画面になる. ほとんど直感的に使用できるため, 1度使ってみてはと思います. 一例として, 曲線の長さを測る場合を示します.
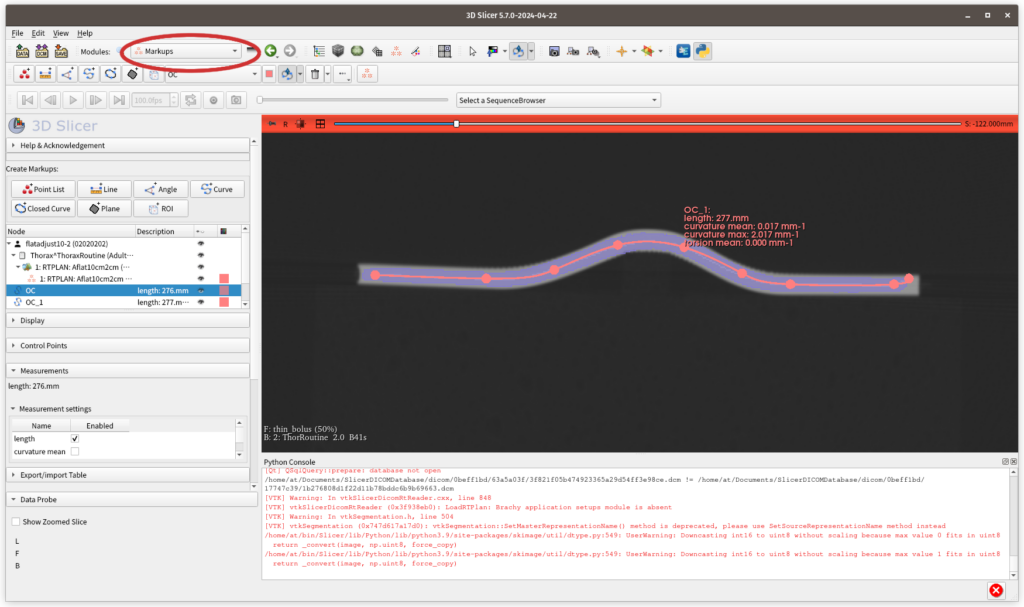
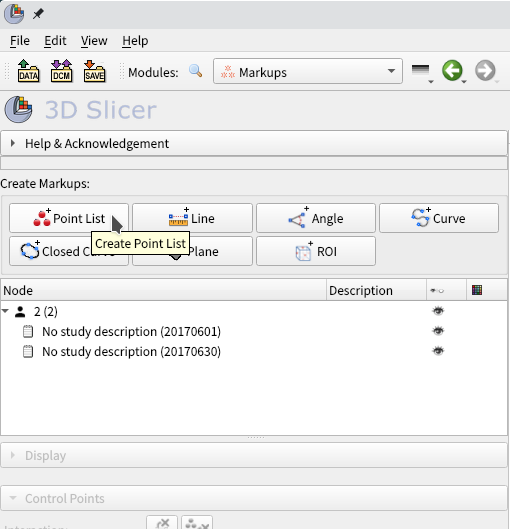
まず, 上記画面のようにModuleのリストからmarkupsを選択して, markupsの画面(左画面)を出します.
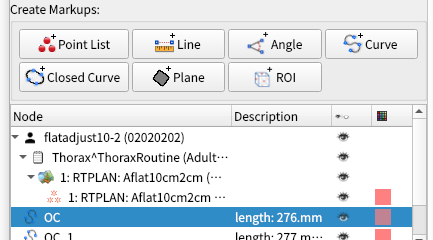
上記ボタンが出ていなければ, [Create Markups]の左にある黒三角をクリックすれば表示されるはずです.この左画面のいくつかのボタンはそれぞれポイント(point list), 直線(Line), 角度(Angle), 曲線(Curve), 閉曲線(Closed Curve), 面(Plane), 関心領域(ROI)を作るボタンになります. 使いたいものを作って(描いて)それを計測に使うことになります.
今回曲線の距離を測るので, [Curve]を選択しました. ボタン1回押すごとに, 上記画像の下部にある[OC]という項目が増えていきますので注意してください. このOCが今回計測するための曲線になります.

次に画像が表示されている画面上で点を取っていきます. このあたりはどのようなソフトでも同様です. 最後はダブルクリックで終点が設定されます.
上記画面ではすでに曲線に関する数値が出ていますが, それらの表示項目を設定するのが, 次の[Measurements]の設定になります. したの画像のような画面が出ていなかれば[Measurements]横の黒三角をクリックするとこで表示されます.
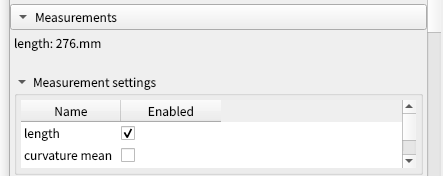
Measurement settingにある項目は, 選択したmarkupの種類, 今回は[Curve]を選択したので, それに合わせて測定できる項目が変わります. [Curve]には, length, curvature mean, curvature max, torsion mean, torsion maxが選択できます. ここでチェックをつけた項目が[Measurement setting]の上の部分に数値が表示されます. また,画像上でも表示されることになります.
今回の例では, たわんだボーラスの長さを計測していますが, 結果は276 mmとなっています.
計測した数値は自身でメモる必要があります. [Measurements]の少ししたに[Export/Import Table]という項目があるので結果を表に出せそうに見えますが, こちらはmarkupを形成するポイントの情報を表示出力してくれるだけになります. それらは[Control points]で見れる情報と同じです. つまり,markup自体を外部保存したり取り込んだりするための機能のようです.
これら計測は直感的ですので, その他の設定項目やmarkupの種類など自身で試して確認してみてください.