Yoichi Watanabe, N. Gopishankar
Journal of Applied clinical Medical Physics, Vol. 12, no. 1, Winter 2011
DOI: 10.1120/jacmp.v12i1.3273Citations: 9
(日本語)
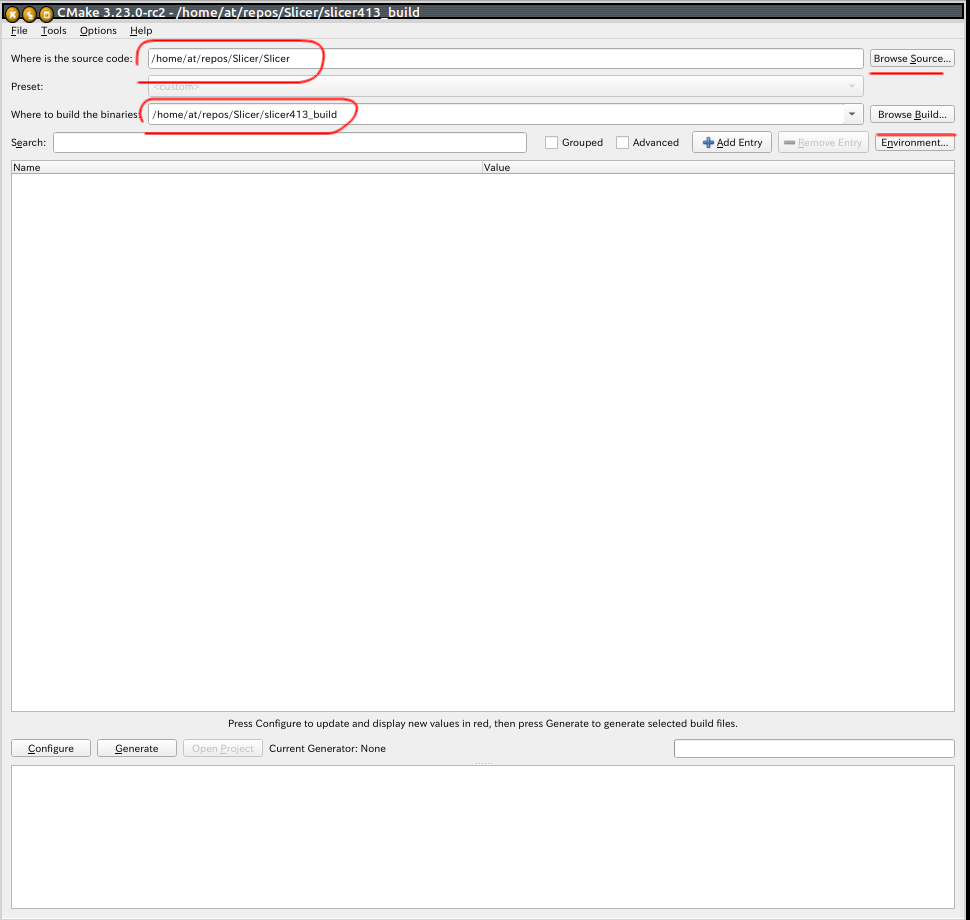
放射線治療において,線量計算や治療計画に使用するソフトウェアの検証は,正確な放射線照射を行うために非常に重要である.BANG3ポリマーゲル線量計と3T Magnetic Resonance Imaging(MRI)を用いて,トモセラピー治療計画および放射線照射の精度検証を行なった.前立腺治療1症例を評価し、計算された3次元線量分布は,最大線量(2.5Gy)よりはるかに小さい(25%以下)領域を除いて,測定された3次元線量分布と一致することが分かった。計画標的体積を含む12cm×11cm×9cmのボリュームに対してγIndex(3%/3mm)を用いた解析では,53%のボクセルでγIndexが1より小さいことが示された.この測定プロトコルと解析ツールは、強度変調回転放射線治療のような他の新しい 複雑な放射線照射技術の評価にも,低コストかつ容易に適用することが可能である。