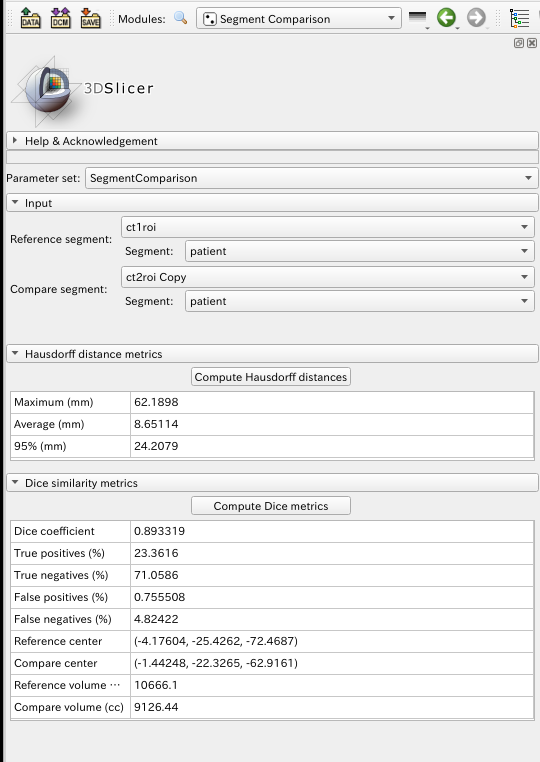
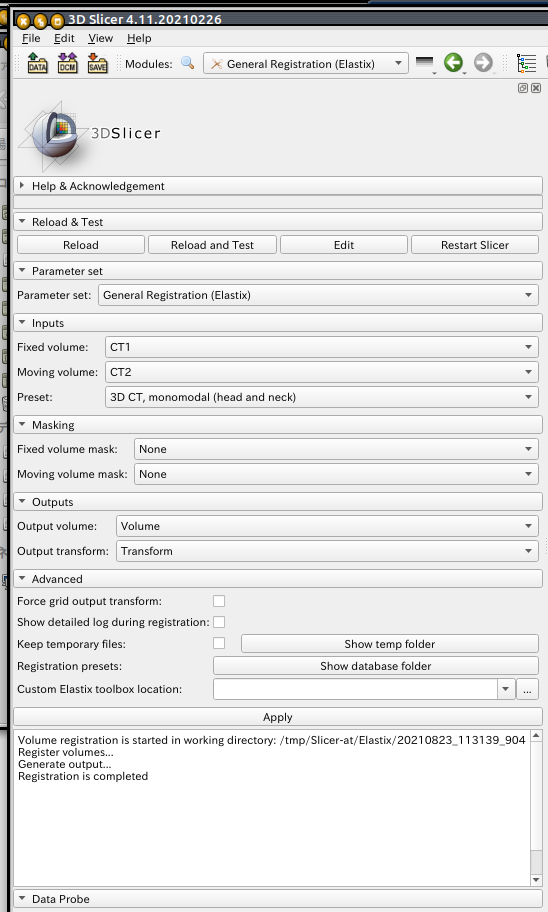
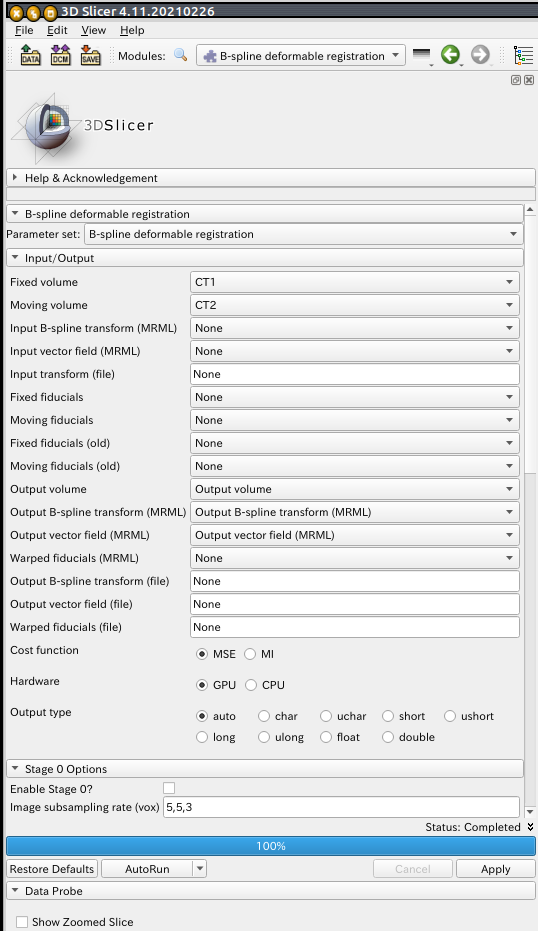
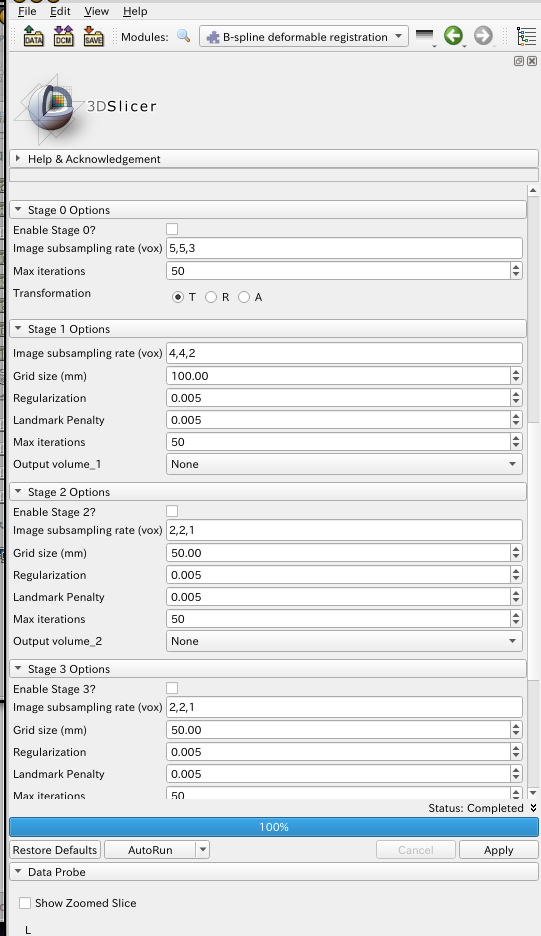
OS: Ubuntu 20.04, 3DSlicer Version: 4.11.20210226
3D Slicerを用いてJacobian Determinant (jacobian)画像 およびInverse Consistency (ICE) 画像を計算する方法を記載します.
それぞれの画像から値を取る方法はまた別に記載します.
また, DIRの評価ですので, DIRがすでに終えている状態からの処理になります.
前準備
JacobianやICEを計算するために, RegistrationQA Extensionを使用します. 事前にExtension Managerからインストールしておいてください. 画面をキャプチャした時がたまたまなのかわかりませんが, アイコン画像が表示されませんでした(下のような感じです).
前準備2つ目は, DIRで得られたDVF (3D SlicerでDIRした場合TransformNodeでも可)データをRegistrationQAの計算に使えるよう, VectorVolumeデータに変換します.
まずはDIR後のデータを一覧します. ReferenceをCT1, MovingをCT2としたDIRが順方向とし, deformedCT2が変更後のCT画像, ???_goとなっているものがDVFデータです. ICEを計算するのに逆方向も必要ですので逆方向で変形したdeformedCT1が変形画像, ???_returnとついているものがDVFデータです.
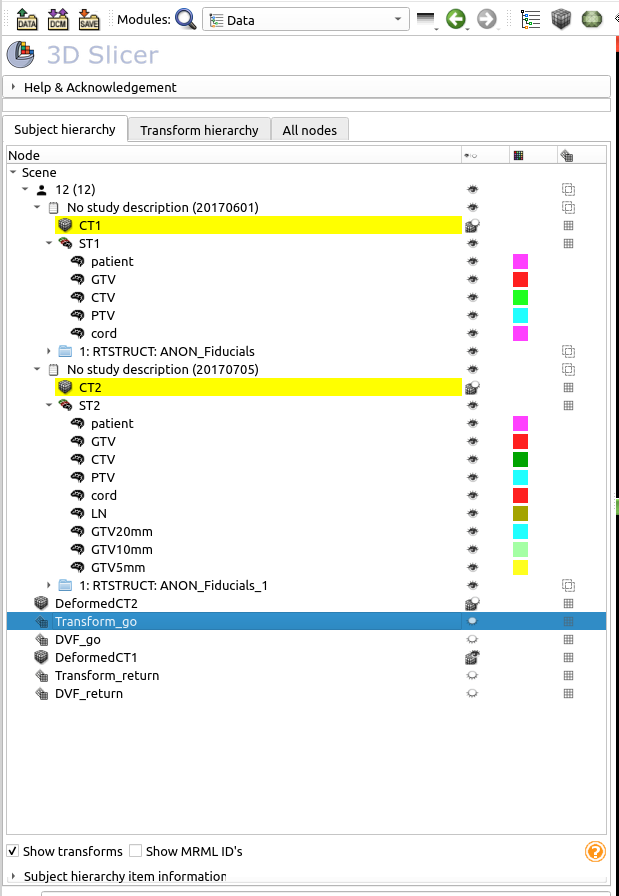
順方向のDVFデータ, Transform_goと DVF_goどちらでもいいのですが, 今回名前の短く, モジュール名と重ならないDVF_goを使います.逆方向も同様です.
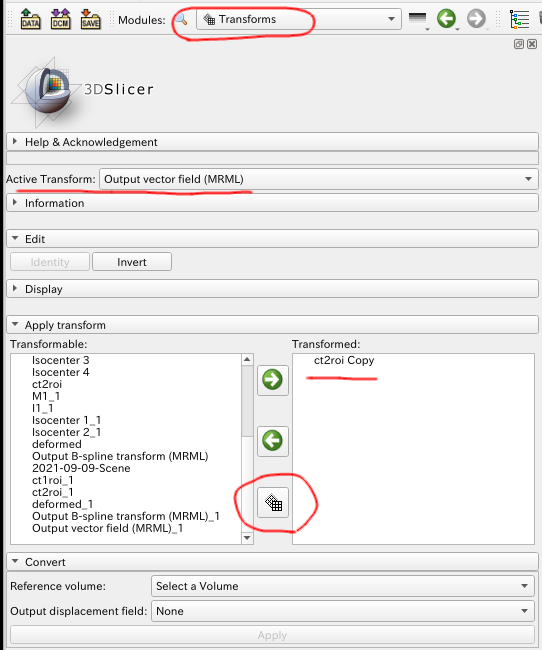
DVF_go, およびDVF_returnをTransformデータからVectorVolumeデータに変換します. 変換はTransformモジュールを使用します.
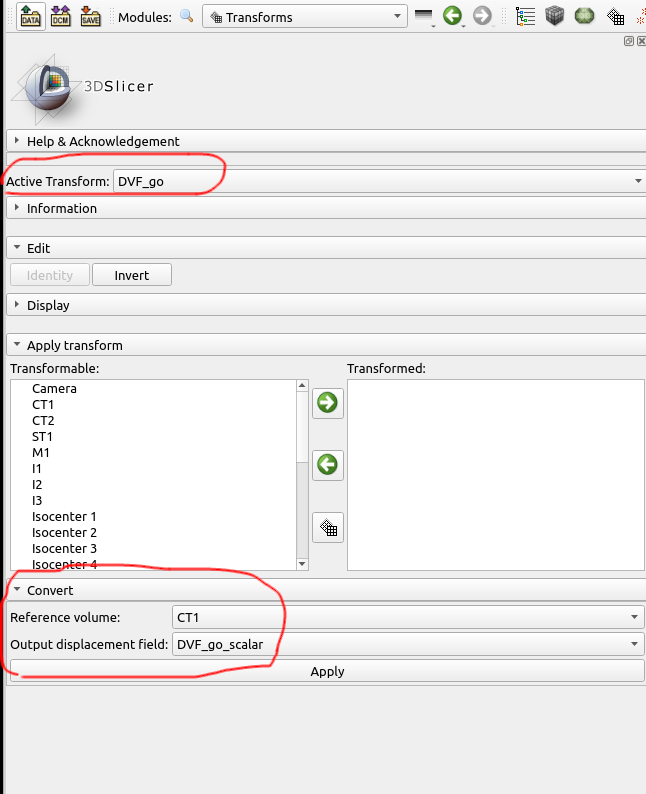
Transformモジュールに切り替えると, 下のようなパネルになります. その中で「Active Transform:」(図中上部赤丸)に変換したいDVFデータを指定します. 下の例ではDVF_goが選択されています. 逆方向の場合はここはDVF_returnとなります.
次に間の項目は無視して, Convertの見出しまで飛びます. Convertの設定項目で, Reference Volumeとoutput displacement fieldを入力します.
Reference Volumeは先に指定したDVFを得たときの(DIRしたときの)Reference画像を指定します. ここでは順方向のReference画像はCT1ですので, CT1を指定しています.
Output Displacement Fieldは変換後のデータ名となります. ここで注意ですが, 出力できるデータ形式は次の3つがあります.
Transformデータ(もとのDVF_goの形式と同じ), Volumeデータ, VectorVolumeデータ
必ず「Create new VectorVolume」もしくは「Create new VecorVolume as…」を選んでVectorVolumeに変換するようにしてください. 下の例では「Create new VecorVolume as…」を選んでDVF_go_scalarと言う名前になっています.
以上を設定したら, 後はApplyボタンを押すだけです. これも変換にしばらく時間がかかりますので気長に待ってください.
Jacobian, ICEの計算
モジュールをRegistrationQAに切り替えます. モジュールの中の「Registration」の中に「Registration Quality Assurance」があるので, 選択してください. 事前にRegistrationQA Extensionを入れていればあると思います.
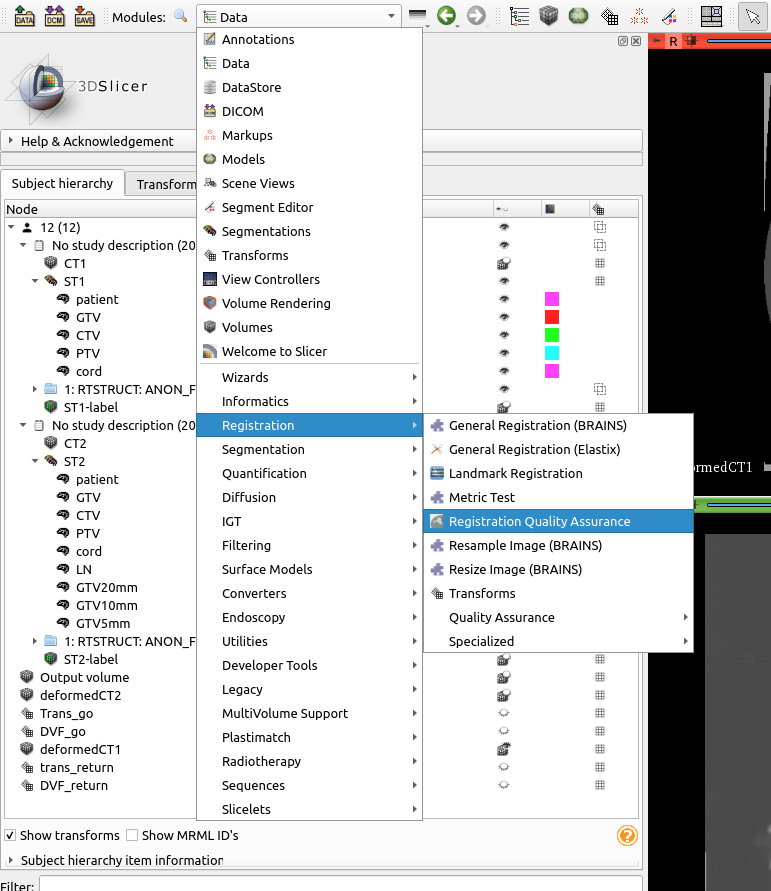
下がRegistration Quality Assuranceを選択した際のパネルです. 今回使うのは今まだ非アクティブですが, 下の方の「Jacobian Determinant」と「Inverse Consistency」ボタンです. 必要なデータが登録されるとアクティブになります.
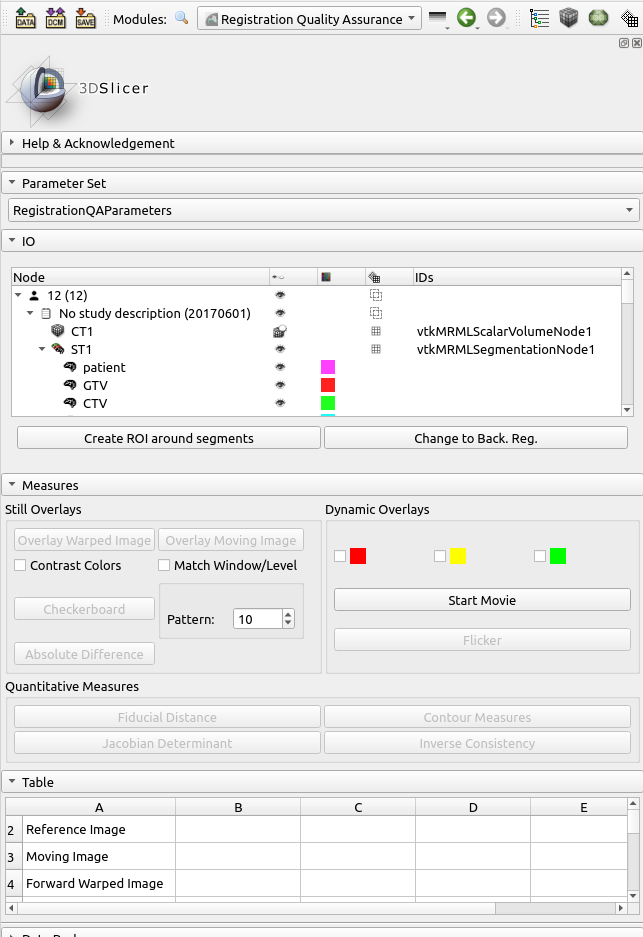
次に, 使用するデータを登録していきます. この方法が直感的でなく, わからなくなるところです.
使用するデータを登録するにはパネル上部にある「Node」の一覧表で右クリックして出てくるメニューで「RegistrationQA」→「Assign node to:」と選んでいき, それぞれ適切なデータを適切な種類にassignしなくてはなりません.
下の画像では, CT1をReference Imageであるとして登録しました. その他Image系では, Moving image, Forward warped Image, Backward warped imageを登録します. 今回順方向ではCT2をmovingにしましたので, 同様な方法でCT2をMoving imageに登録しました.
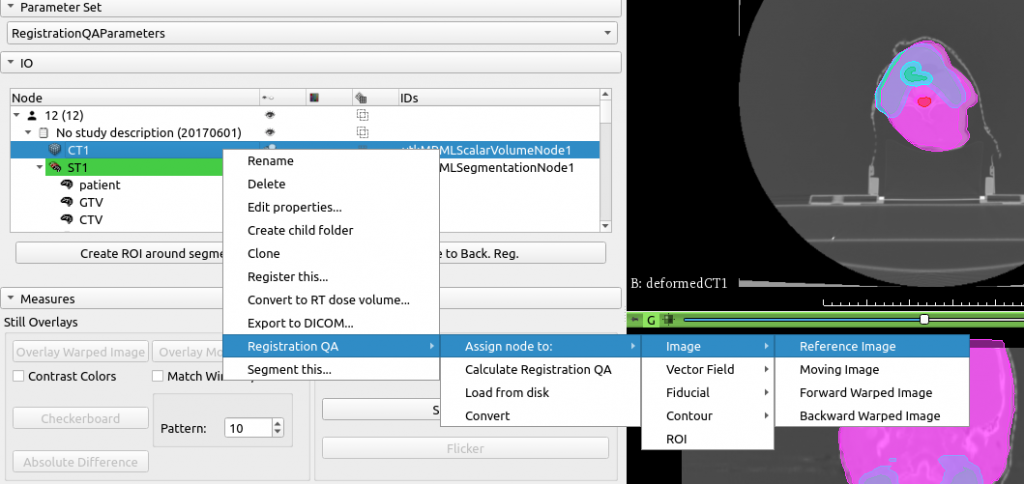
Forward warped imageは順方向での変形後の画像,Backward warped imageは逆方向での変形後の画像を指定すると思われます. Reference, Movingは順方向(もしくは逆方向)でのセットなのに, 変形後の画像は順方向, 逆方向両方を登録できることになります.
さらに 混乱を招くところですが, パネル一番上の項目にParameter Setというのがありますが, その下のプルダウン(下の図では「RegistrationQAParameters」を広げると「RegistrationQAParameters_Backword」というのが選択しにあります. おそらく, ReferenceとMovingを逆にして登録すればいいと思いますが, 再度変形後の画像を登録するのか, 登録知るのならどっちがforwardになるのか, よくわかりません.
Vector FieldへDVFを登録する際ですが, 方法はCTと変わらず登録するデータを一覧表から選んで右クリックし, 「Assign node to」で「Vector Field」を選んで登録します. Vector Fieldには「Forward」と「Backward」があり(!?) それぞれに, 前準備で変換したVectorVolumeのデータを登録します. Transformデータも登録できてしまうのですが, 計算はされません(しかもなんのエラーも出ません).
今回のようにJacobianとICEを求める場合は, Vector Fieldをassignすれば計算できます. CT画像も必要ありません. JacobianはForwardかBackwardのどちらかさえ登録すればアクティブになります.
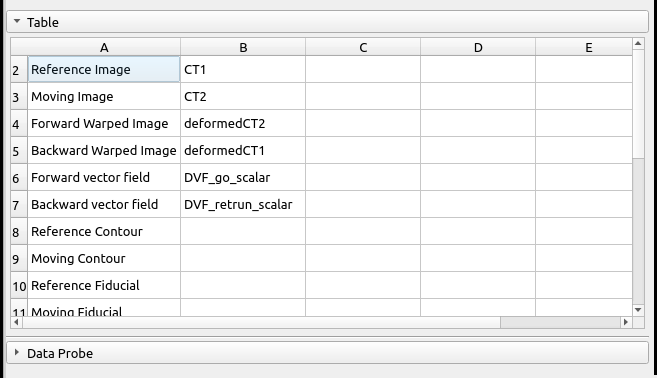
データ登録がされると, パネル下方のTableの項目にAssignしたデータ名が表示されます. この表の続きには, JacobianやICEを計算したあとにそれぞれの値(平均や最大, 最小など)が入る項目もあります.
必要なデータの登録がすんだら「Jacobian Determinant」「Inverse Consistency」ボタンが有効になっているはずですので, それぞれをクリックして計算させてください. 一つ一つ計算させることになりますが, この計算もまた少し時間がかかります. 計算が終わるとそれぞれ計算された画像が表示されます. (下図はJacobianを表示)
また, Tableの中に「Jacobian (for)」と「Jacobian (Back)」があり, その並びに数値が入っていると思います.
さらに, データ一覧(パネル上部のIOの表) に, Vector FeildにassignしたDVFデータの尾末に 「-jacobian」や「-invConsist」とついたデータが出来ていると思います. これでJacobianやICEのデータが画像データとして作成されたことになります.
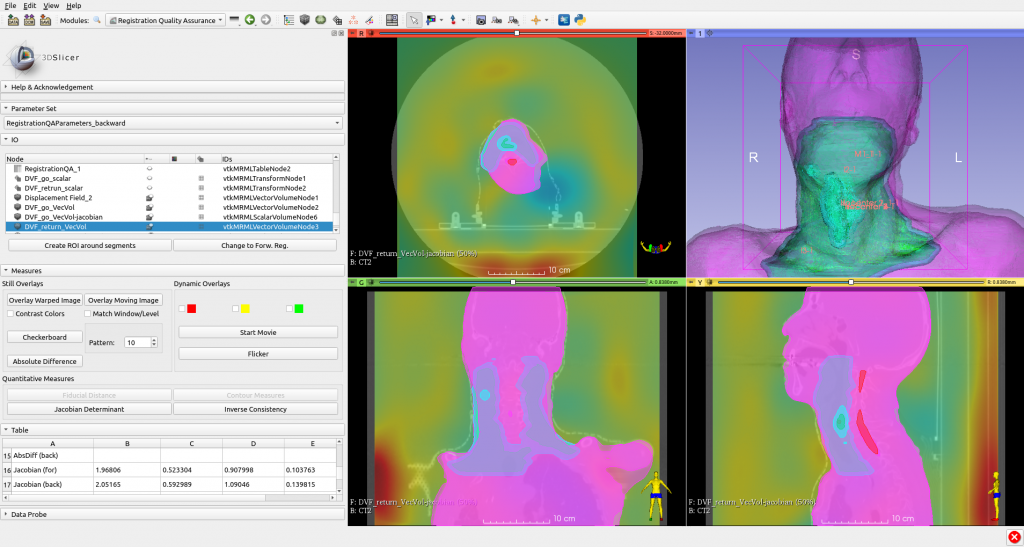
ここまでで, JacobianとICEを画像データとして作成できました. ただし, まだストラクチャに対して個別にそれぞれの値をとることが出来ていません. 次の投稿ではSegment statisticモジュールを使って数値を読む方法を記載します.