OS: Ubuntu 20.04, 3DSlicer Version: 4.11.20210226
3D Slicerを用いると, (たぶん読み込める画像なら)どのような画像でもDICOM形式に変換できます.
ここでは, DICOM化の方法を記載しておきます.
Fileの読み込み
基本的に何でもいいのですが, JPEGなどであると1スライスの画像とみなされます. 試していませんが, ファイルを開く際にフォルダを指定できるので, フォルダを指定すると3Dデータになるかもしれません.
今回は本当に一般的な画像がDICOM化できることを示すためにJPEGでやってみました.
3DSlicerのメニューの「File」から「Add Data」を選ぶか, メニューの下の一番左のDataアイコンをクリックすると下のウィンドウが出てきます. 今回, JPEGファイル1つなので「Choose File(s) to Add」を選んでファイルを選択しました. 「Choose File(s) to Add」をクリックするとさらにファイル選択のウィンドウが出てきます.
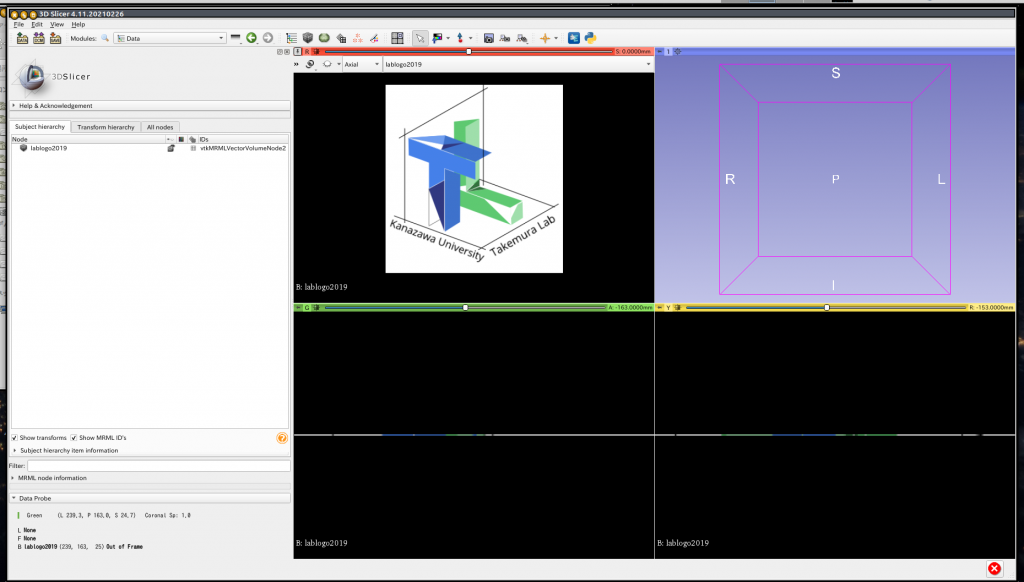
ファイルを選択し, 開くと下の図のような画面です. ファイルは研究室のロゴマークのファイルにしました. 1枚の2D画像ですので, Sag, Corの画像は線です.
DICOM化
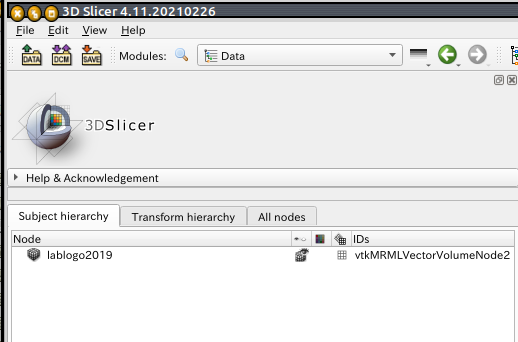
もう早速DIROM化です. まず下図のように, Modules横のプルダウンを「Data」にしてください. そうすると左パネルに「Subject hierarchy」タブになっていると思います(なっていなければ選択してください).
DICOMーRTなどを開くと輪郭などがツリー状に表示されますが, これまでの手順でファイルを開くと開いたファイルのファイル名が1つだけ表示されていると思います. (しっかりCT画像などと同じvtkMRMLVolumeNodeの1種になっているのがわかります)
次に, ファイル名を選択し右クリックします. 出てきたメニューに「Export to DICOM…」という項目があるので選択します.
そうすると下の警告が出ます.
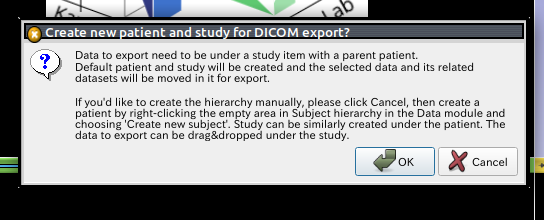
警告に書いているとおりですが, DICOM(化)には親となる患者情報が必要でOKを選ぶとデフォルトでつくられる. もし自分で作りたければ一度Cancelしてsubject hierarchyのところで, 「Export to DICOM…」を選んだメニューから「Create new subject」を選んで作成してくださいとのことです. Subjectを作った後にsubject hierarchy上でファイル名をドラッグ&ドロップすれば,作成したSubjectの配下に移動できます.
DICOMの情報は次のところで編集ができるのでそのままOKで問題ないと思います.
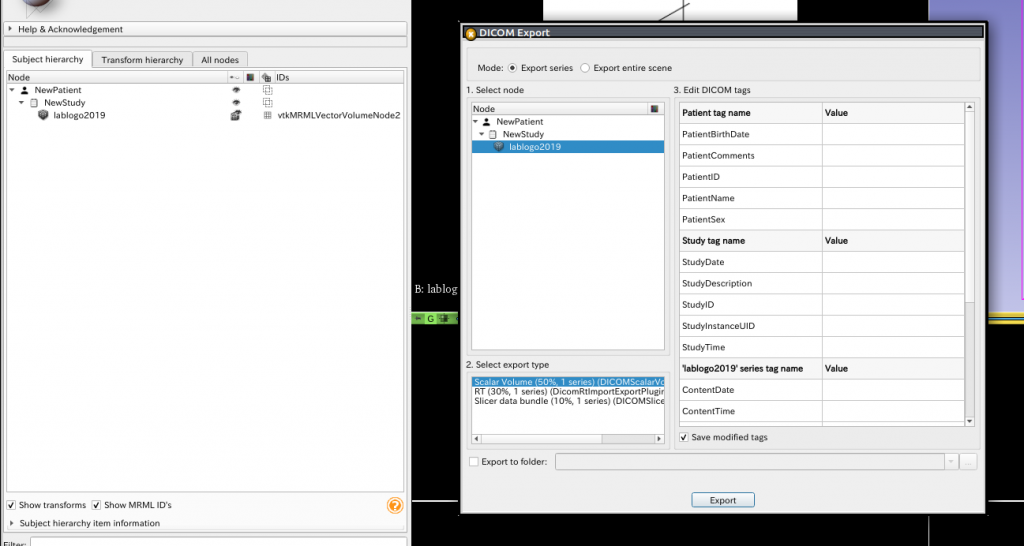
そのままOKすると下図右のようなウィンドウが出ます. ここでDICOMのヘッダに含めるMeta情報を入力していきます. とりあえずDICOM化したいだけであれば, なにも変更しなくてもDICOM化できます.
ついでに, 下図左のsubject hierarchyのパネルには, 自動にNewPatientとNewStudyが出来てその下にロゴファイルの名前が移動していると思います.
DICOM化したデータをそのまま3DSlicerのDICOMモジュールが管理するようにしたければ(3DSlicerのDICOM画像管理画面で出てくるようにしたければ), 「Export」をクリックしてください. DICOM画像のリストに適当につけられた患者名となってあるのが確認できます.
ファイルとして出力したい場合は, 下図右のウィンドウ下部の「Export to folder」にチェックを入れて, その右に出力先のフォルダを指定してください.
3DSlicerのDICOM管理に出力した場合, 以降は通常のDICOM画像と同様なので, もしファイルとしてExportしたければDICOMの管理画面で可能となります.
確認
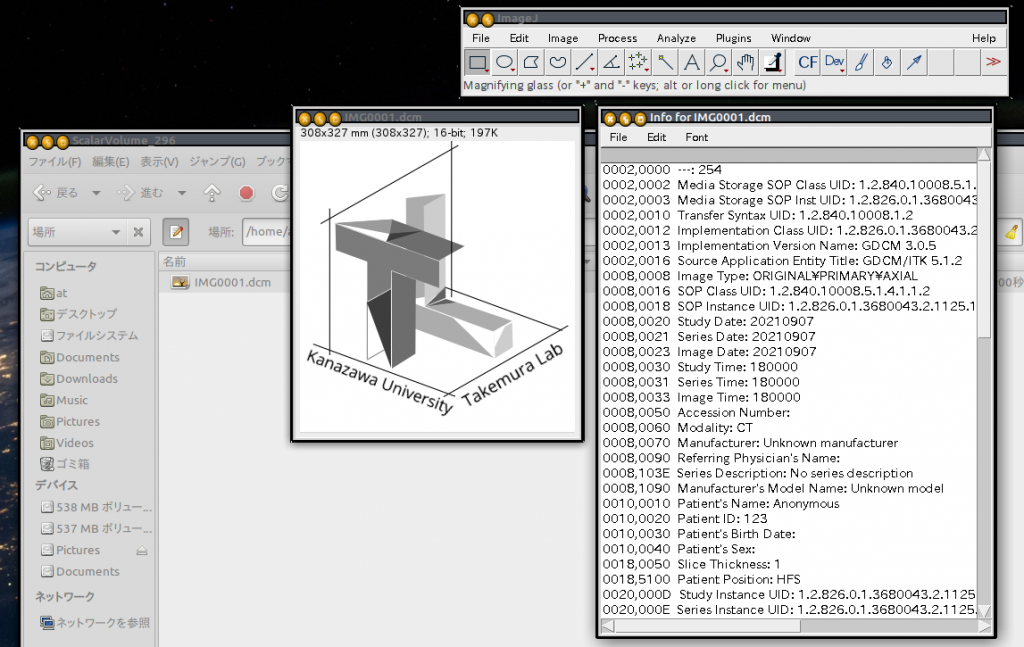
最後にちゃんと出来たか確認ですが, ImageJを使って確認してみました. Exportしたファイルは1枚の画像ですので指定したフォルダには「IMG001.dcm」というファイルが出来ていました. そのファイルをImageJにドラッグ&ドロップすると白黒化されたロゴが表示されました. DICOMですのでグレーレベルの画像になったようです. ちゃんとDICOMかどうか確認するため, Image infoを表示させると, ちゃんとタグとともにタグ情報も表示されました.
さらにちゃんとしているのがUIDがついていること. 正解で一つしかない一意な番号がつけられるUIDをどのように決めているのか少し謎です. とはいえ, ちゃんとDICOM化が出来ました.